Ando com o sono agitado ultimamente. Em janeiro de 2024 – há quase dois anos, comecei a pensar em uma estrutura ou modelo de dados para “plantas medicinais”, provocado pela querida amiga, Dra Viviane Kruel. A proposta era ousada! Passar o nome vernacular para o protagonista do modelo, em substituição ao nome científico. De lá para cá, como podem ver até pelas postagens aqui no blog, meu envolvimento com alunos de pós-graduação e iniciativas relacionadas com o tema da etnobotânica se intensificou significativamente. Da Rede de Conhecimentos da Sociobiodiversidade, tocada pelo ICMBio ao projeto para o “Desenvolvimento de uma extensão de dados para documentar informações sobre a sociobiodiversidade“, com o SiBBr e parceiros. Do projeto MCTI/GEF “Empowering Indigenous Peoples and Local Communities (IPLCs) to manage biodiversity data and information as a strategy to conserve their territories, safeguard traditional knowledge, and promote integrated biodiversity management” à iniciativa USEFLORA, e ainda considerando a parceria da RNP para o desenvolvimento de um sistema para o SisGen, a interseção do interesse destas e outras iniciativas relacionadas e equivalentes é a materialização de um “banco de dados sobre conhecimento tradicional”.

Cheguei a apresentar uma proposta de “Arquitetura para um Sistema de Informações sobre Conhecimento Tradicional Associado à Biodiversidade“, na Oficina da Rede de Conhecimentos sobre a Sociobiodiversidade, no início de dezembro de 2025, mas o que vem tirando meu sono é a “camada de persistência” desta arquitetura. O banco de dados em si. Mais precisamente, as visões e implementações de modelos de dados válidos.
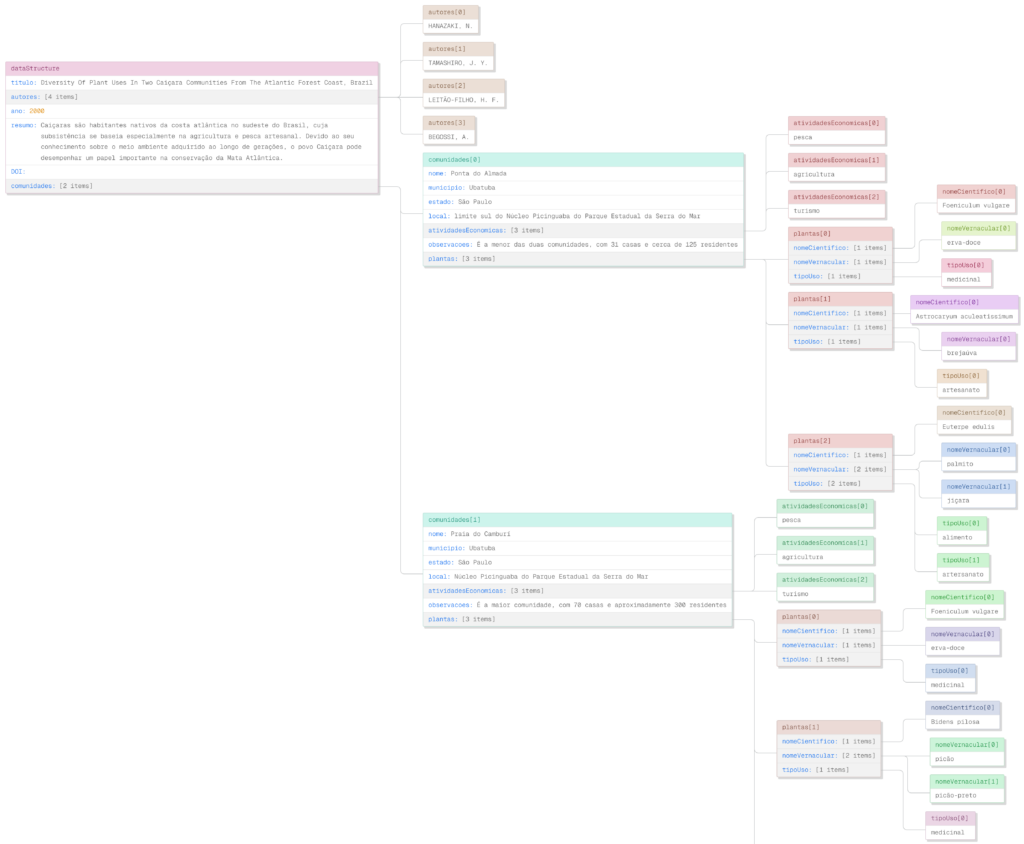
Semana passada estive na oficina de trabalho do projeto USEFLORA, onde uma visão que considero amadurecida e robusta brotou, como o propósito de um banco de dados de conhecimento tradicional baseado em dados secundários: “a relação das comunidades tradicionais com as plantas, evidenciada por artigos científicos publicados”. Simples, mas preciso. O pessoal com a cabeça no modelo relacional já visualizou pelo menos três entidades: comunidades, plantas e referências. Nenhum problema ai.
Porém, minha cabeça me levou para um modelo de grafo, onde se a referência bibliográfica é o testemunho factual da relação entre uma comunidade e um conjunto de plantas, deve ser representado como uma “relação” (edge) em um modelo baseado em grafos!
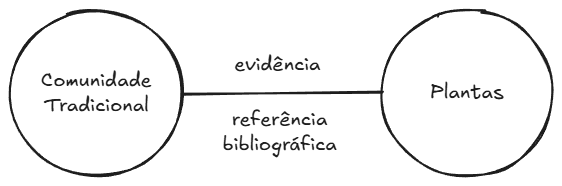
Neste caso, temos dois “nós” (nodes), comunidades e plantas; e um “relacionamento” (edge), que guarda as propriedades da referência que cita a relação entre uma comunidade e um conjunto de plantas. É ai que a teoria – elegante, em minha opinião – começa a fraquejar frente à implementação prática da solução.
Como já tenho alguma experiência com alguns bancos de dados orientados à grafos, parti para o teste da implementação. Primeiro, vale comentar que deixei de lado soluções muito “imaturas”, apesar de muito promissoras, como o SurrealDB, já comentado aqui. Passada esta tentação, parti para o BD orientado a grafos mais utilizado e maduro – Neo4j. Com o Neo4j, que hoje o Neo4j tem uma versão desktop bem amigável, criei um BD com meia duzia de registros, para reflexão.
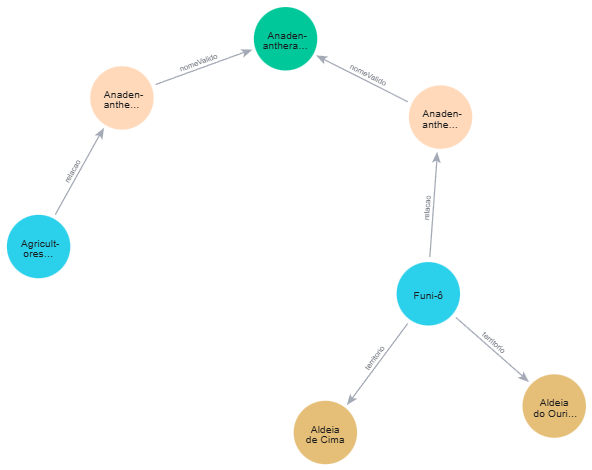
Ai já vem o primeiro “choque de realidade”: a funcionalidade de “chave-valor” para armazenar as propriedades dos nós e relações, associados às “etiquetas” (labels) é inadequado para guardar (e recuperar!) propriedades complexas das comunidades, das plantas e das referências. De volta à prancheta!
Meu segundo teste foi com o ArangoDB. Gosto muito desta ferramenta, “meio” multi-modelo (não tão quanto o SurrealDB), mas com a capacidade de usar documentos JSON como registro de nós e relações! Show! Limitação da “chave-valor” do Neo4j resolvida, e considerando que a “community edition” (gratuita) seria suficiente para algumas das iniciativas (100GB). Vamos à implementação.
Enquanto vou entrando registros, vou pensando nas limitações da implementação em um abiente de produção como, por exemplo, aprender “Arango Query Language” (AQL), e que uma referência que relatou a relação de uma comunidade com “n” plantas, vai gerar uma redundância de “edges” bem significativa. Entre outras coisas… Afinal, não existe modelo nem banco de dados perfeito!
Alguns dias mal dormidos depois, e alguns milhares de créditos com o Claude “queimados”, convenci-me de que a elegância deveria dar lugar à praticidade. Partindo de um modelo bem estruturado em JSON, seria possível ter uma base de dados em MongoDB que representasse a realidade do documento-testemunho – o artigo científico – sem precisar adotar uma estrutura de grafos. Afinal, sequer sabemos se algoritmos e métricas de rede serão necessários para analisar e expor estes documentos.
Um documento JSON é generoso o suficiente para registrar toda a heterogeneidade não só dos artigos (que podem ainda ser adaptados para livros, relatórios etc.), mas das comunidades e das relações com as plantas. A falta de um protocolo unificado ou acordado que permeia as pesquisas acadêmicas sobre o tema, gerou – e vem gerando – artigos científicos completamente heterogêneos nos dados e informações que contém, tanto das plantas quando das comunidades. Assim, um banco de dados tido como “schemaless” parece ser muito mais adequado para uma realidade como esta.
Claro que o “Sr. ansioso” aqui tinha que partir para a emplementação de uma interface, nos contextos de aquisição, curadoria e apresentação de dados, conforme visualizado na arquitetura proposta e publicada. E mais um repositório nasceu: etnoDB – Base de Dados Etnobotânica. Ainda engatinhando, mas pretende ser uma “prova de conceito” da proposta da arquitetura implementada sobre um banco de dados NoSQL, orientado a documentos JSON (MongoDB).
Comentários, críticas e sugestões são sempre bem vindos!